Laboratoire Epigénétique et destin cellulaire (EDC) :
Equipe DNA méthylation and ncRNA in health and disease
http://parisepigenetics.com/dmrhd/
http://parisepigenetics.com/dmrhd/
Florent HUBE, Baptiste BORGARD, Lucile GUICHARD
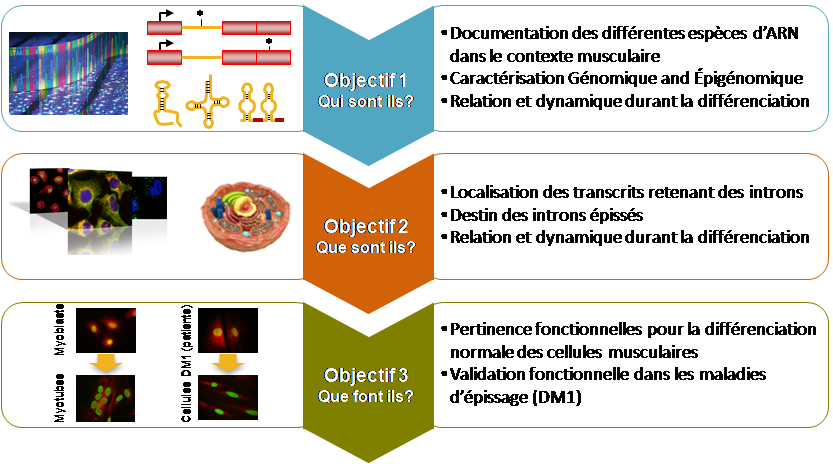
ARNnc, Introns, Dystrophie musclaire 1 (DM1)
Le génome humain est composé de seulement 2% de séquences codantes et de plus de 50% d’introns. Les introns sont transcrits et généralement dégradés. Néanmoins, les introns contiennent des informations génétiques importantes ; ils peuvent être les précurseurs de petits ARN régulateurs tels que les microARN et les petits ARN nucléolaires (snoARN), tous impliqués dans la régulation de l’expression des gènes au niveau post-transcriptionnel. Comme l’épissage de l’ARN est une étape critique pour l’expression des gènes chez les eucaryotes, toute perturbation entraînera des maladies humaines comme la dystrophie myotonique de type 1 (DM1). Pourtant, l’épissage alternatif des introns reste mal documenté dans ces contextes. Récemment, nous avons mis en place un RNA-seq original sur ARN fractionné en taille dédié à l’identification systématique des SID (Short Intron-Derived ncRNA), sans a priori sur leur biogenèse et leur origine génomique, dans le contexte des cellules satellites normales et DM1. Nous avons identifié plusieurs classes de SID qui sont exprimées de manière différentielle lors de la différenciation musculaire normale et dont les niveaux sont remarquablement perturbés dans les cellules DM1. Les candidats les plus perturbés appartiennent à la classe des snoARN. Nous avons découvert de nouveaux membres d’une famille de snoARN (snoX) dont les niveaux d’expression augmentent pendant la différenciation musculaire normale mais sont drastiquement réduits dans les cellules DM1. Des expériences de perte et de gain de fonction révèlent que la famille snoX est impliquée dans la différenciation musculaire. En effet, des niveaux réduits de snoX altèrent la fusion cellulaire dans les cellules normales alors que la surexpression de snoX favorise la fusion cellulaire dans les cellules DM1.
[1] Bogard B, Francastel C, Hubé F. Multiple information carried by RNAs: total eclipse or a light at the end of the tunnel? RNA Biol. 2020; 17(12):1707-1720. PMID: 32559119; PMCID: PMC7714488.
[2] Hubé F, Ulveling D, Sureau A, Forveille S, Francastel C. Short intron-derived ncRNAs. Nucleic Acids Res. 2017; 45(8):4768-4781. PMID: 28053119; PMCID: PMC5416886.
[3] Hubé F, Francastel C. « Pocket-sized RNA-Seq »: A Method to Capture New Mature microRNA Produced from a Genomic Region of Interest. Noncoding RNA. 2015; 1(2):127-138. PMID: 29861419; PMCID: PMC5932543.
[4] Ulveling D, Dinger ME, Francastel C, Hubé F. Identification of a dinucleotide signature that discriminates coding from non-coding long RNAs. Front Genet. 2014; 5:316. PMID: 25250049; PMCID: PMC4158813.